Keyword: マハラノビス, 二乗距離, 計算, 多変量解析
概要
本サンプルはマハラノビス二乗距離の計算を行うC言語によるサンプルプログラムです。 本サンプルは以下に示されるデータについてマハラノビス二乗距離の計算を行います。
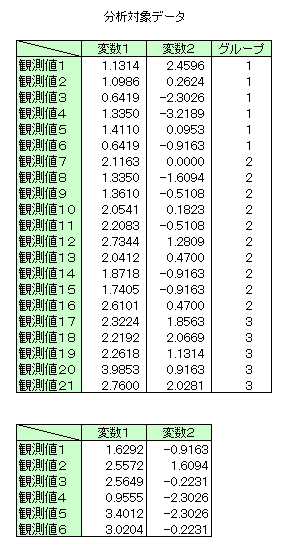
※本サンプルはnAG Cライブラリに含まれる関数 nag_mv_discrim_mahaldist() のExampleコードです。本サンプル及び関数の詳細情報は nag_mv_discrim_mahaldist のマニュアルページをご参照ください。
ご相談やお問い合わせはこちらまで
入力データ
(本関数の詳細はnag_mv_discrim_mahaldist のマニュアルページを参照)| このデータをダウンロード |
nag_mv_discrim_mahaldist (g03dbc) Example Program Data 21 2 2 3 Nag_FALSE 1.1314 2.4596 1 1.0986 0.2624 1 0.6419 -2.3026 1 1.3350 -3.2189 1 1.4110 0.0953 1 0.6419 -0.9163 1 2.1163 0.0000 2 1.3350 -1.6094 2 1.3610 -0.5108 2 2.0541 0.1823 2 2.2083 -0.5108 2 2.7344 1.2809 2 2.0412 0.4700 2 1.8718 -0.9163 2 1.7405 -0.9163 2 2.6101 0.4700 2 2.3224 1.8563 3 2.2192 2.0669 3 2.2618 1.1314 3 3.9853 0.9163 3 2.7600 2.0281 3 1 1 6 Nag_NotEqualCovar 1.6292 -0.9163 2.5572 1.6094 2.5649 -0.2231 0.9555 -2.3026 3.4012 -2.3026 3.0204 -0.2231
- 1行目はタイトル行で読み飛ばされます。
- 2行目に観測値の数(n)、変数の数(m)、分散共分散行列の変数の数(nvar)、グループの数(ng)、重みづけをするかどうか(weight)を指定しています。 "Nag_FALSE"は重みづけをしないことを意味します。
- 3〜23行目に変数の観測値(x)と観測値がどのグループに属するか(ing)を指定しています。
- 24行目に変数が距離計算に含まれるかどうか(isx)を指定しています。"1"の場合は計算に含まれます。
- 25行目に割り当てられる観測値の数(nobs)、グループ内の分散共分散行列が等しいと仮定されるかどうかを示すパラメータ(equal)を指定しています。"Nag_NotEqualCovar "はグループ内の分散共分散行列が等しくないことを意味します。
- 26〜31行目に変数の新たな観測値(x)を指定しています。
出力結果
(本関数の詳細はnag_mv_discrim_mahaldist のマニュアルページを参照)| この出力例をダウンロード |
nag_mv_discrim_mahaldist (g03dbc) Example Program Results Obs Distances 1 3.339 0.752 50.928 2 20.777 5.656 0.060 3 21.363 4.841 19.498 4 0.718 6.280 124.732 5 55.000 88.860 71.785 6 36.170 15.785 15.749
- 4〜12行目に何番目の観測値か、マハラノビス二乗距離が出力されています。
ソースコード
(本関数の詳細はnag_mv_discrim_mahaldist のマニュアルページを参照)
※本サンプルソースコードはnAG数値計算ライブラリ(Windows, Linux, MAC等に対応)の関数を呼び出します。
サンプルのコンパイル及び実行方法
| このソースコードをダウンロード |
/* nag_mv_discrim_mahaldist (g03dbc) Example Program.
*
* CLL6I261D/CLL6I261DL Version.
*
* Copyright 2017 Numerical Algorithms Group.
*
* Mark 26.1, 2017.
*
*/
#include <nag.h>
#include <stdio.h>
#include <nag_stdlib.h>
#include <nagg03.h>
#define D(I, J) d[(I) *tdd + J]
#define X(I, J) x[(I) *tdx + J]
int main(void)
{
Integer i, j, m, n, ng, nobs, nvar, tdd, tdgmean, tdx;
Integer exit_status = 0, *ing = 0, *isx = 0, *nig = 0;
char nag_enum_arg[40];
double df, sig, stat;
double *d = 0, *det = 0, *gc = 0, *gmean = 0, *wt = 0;
double *wtptr = 0, *x = 0;
Nag_GroupCovars equal;
Nag_Boolean weight;
NagError fail;
INIT_FAIL(fail);
printf("nag_mv_discrim_mahaldist (g03dbc) Example Program Results\n\n");
/* Skip headings in data file */
scanf("%*[^\n]");
scanf("%ld", &n);
scanf("%ld", &m);
scanf("%ld", &nvar);
scanf("%ld", &ng);
scanf("%39s", nag_enum_arg);
/* nag_enum_name_to_value (x04nac).
* Converts nAG enum member name to value
*/
weight = (Nag_Boolean) nag_enum_name_to_value(nag_enum_arg);
if (n >= 1 && nvar >= 1 && m >= nvar && ng >= 2) {
if (!(det = nAG_ALLOC(ng, double)) ||
!(gc = nAG_ALLOC((ng + 1) * nvar * (nvar + 1) / 2, double)) ||
!(gmean = nAG_ALLOC(ng * nvar, double)) ||
!(wt = nAG_ALLOC(n, double)) ||
!(x = nAG_ALLOC(n * m, double)) ||
!(ing = nAG_ALLOC(n, Integer)) ||
!(isx = nAG_ALLOC(m, Integer)) || !(nig = nAG_ALLOC(ng, Integer)))
{
printf("Allocation failure\n");
exit_status = -1;
goto END;
}
tdgmean = nvar;
tdx = m;
}
else {
printf("Invalid n or nvar or m or ng.\n");
exit_status = 1;
return exit_status;
}
if (weight) {
for (i = 0; i < n; ++i) {
for (j = 0; j < m; ++j)
scanf("%lf", &X(i, j));
scanf("%ld", &ing[i]);
scanf("%lf", &wt[i]);
}
wtptr = wt;
}
else {
for (i = 0; i < n; ++i) {
for (j = 0; j < m; ++j)
scanf("%lf", &X(i, j));
scanf("%ld", &ing[i]);
}
}
for (j = 0; j < m; ++j)
scanf("%ld", &isx[j]);
/* nag_mv_discrim (g03dac).
* Test for equality of within-group covariance matrices
*/
nag_mv_discrim(n, m, x, tdx, isx, nvar, ing, ng, wtptr, nig,
gmean, tdgmean, det, gc, &stat, &df, &sig, &fail);
if (fail.code != NE_NOERROR) {
printf("Error from nag_mv_discrim (g03dac).\n%s\n", fail.message);
exit_status = 1;
goto END;
}
scanf("%ld", &nobs);
scanf("%39s", nag_enum_arg);
equal = (Nag_GroupCovars) nag_enum_name_to_value(nag_enum_arg);
if (nobs >= 1) {
if (!(d = nAG_ALLOC(nobs * ng, double)))
{
printf("Allocation failure\n");
exit_status = -1;
goto END;
}
tdd = ng;
for (i = 0; i < nobs; ++i) {
for (j = 0; j < m; ++j)
scanf("%lf", &X(i, j));
}
/* nag_mv_discrim_mahaldist (g03dbc).
* Mahalanobis squared distances, following nag_mv_discrim
* (g03dac)
*/
nag_mv_discrim_mahaldist(equal, Nag_SamplePoints, nvar, ng, gmean,
tdgmean, gc, nobs, m, isx, x, tdx, d, tdd,
&fail);
if (fail.code != NE_NOERROR) {
printf("Error from nag_mv_discrim_mahaldist (g03dbc).\n%s\n",
fail.message);
exit_status = 1;
goto END;
}
printf("\n Obs Distances\n\n");
for (i = 0; i < nobs; ++i) {
printf(" %3ld", i + 1);
for (j = 0; j < ng; ++j)
printf("%10.3f", D(i, j));
printf("\n");
}
}
END:
nAG_FREE(d);
nAG_FREE(det);
nAG_FREE(gc);
nAG_FREE(gmean);
nAG_FREE(wt);
nAG_FREE(x);
nAG_FREE(ing);
nAG_FREE(isx);
nAG_FREE(nig);
return exit_status;
}
