Keyword: スチューデントt-copula, 疑似乱数
概要
本サンプルはスチューデントt-copulaから疑似乱数行列の生成を行うFortranによるサンプルプログラムです。 本サンプルは以下に示される共分散行列をもつスチューデントt-copulaから10個の疑似乱数を生成し出力します。
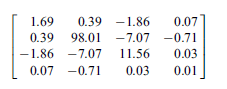
※本サンプルはnAG Fortranライブラリに含まれるルーチン g05rcf() のExampleコードです。本サンプル及びルーチンの詳細情報は g05rcf のマニュアルページをご参照ください。
ご相談やお問い合わせはこちらまで
入力データ
(本ルーチンの詳細はg05rcf のマニュアルページを参照)| このデータをダウンロード |
G05RCF Example Program Data
1 1 1762543 :: GENID,SUBID,SEED(1)
10 4 :: N,M
10 :: DF
1.69 0.39 -1.86 0.07
98.01 -7.07 -0.71
11.56 0.03
0.01 :: End of C (upper triangular part)
- 1行目はタイトル行で読み飛ばされます。
- 2行目に使用する生成器(genid=1:nAG基本生成器)、生成器に関する情報(subid=1:GENIDが1の場合この値は参照されません)、生成器の初期値(seed=1762543)を指定しています。
- 3行目に生成される乱数の数(n=10)と次元の数(m=4)を指定しています。
- 4行目に分布の自由度の数(df=10)を指定しています。
- 5〜8行目に分布の共分散行列の上三角部分(c)を指定しています。
出力結果
(本ルーチンの詳細はg05rcf のマニュアルページを参照)| この出力例をダウンロード |
G05RCF Example Program Results
Variates
1 2 3 4
1 0.6445 0.0527 0.4082 0.8876
2 0.0701 0.1988 0.8471 0.3521
3 0.7988 0.6664 0.2194 0.5541
4 0.8202 0.0492 0.7059 0.9341
5 0.1786 0.5594 0.7810 0.2836
6 0.4920 0.2677 0.3427 0.5169
7 0.4139 0.2978 0.8762 0.7145
8 0.7437 0.9714 0.8931 0.2487
9 0.4971 0.9687 0.8142 0.1965
10 0.6464 0.5304 0.5817 0.4565
- 5〜14行目に生成された疑似乱数が出力されています。
ソースコード
(本ルーチンの詳細はg05rcf のマニュアルページを参照)
※本サンプルソースコードは科学技術・統計計算ライブラリである「nAG Fortranライブラリ」のルーチンを呼び出します。
サンプルのコンパイル及び実行方法
| このソースコードをダウンロード |
PROGRAM g05rcfe
! G05RCF Example Program Text
! Mark 23 Release. nAG Copyright 2011.
! .. Use Statements ..
USE nag_library, ONLY : g05kff, g05rcf, nag_wp, x04caf
! .. Implicit None Statement ..
IMPLICIT NONE
! .. Parameters ..
INTEGER, PARAMETER :: lseed = 1, nin = 5, nout = 6
! .. Local Scalars ..
INTEGER :: df, genid, i, ifail, ldc, ldx, lr, &
lstate, m, mode, n, subid
! .. Local Arrays ..
REAL (KIND=nag_wp), ALLOCATABLE :: c(:,:), r(:), x(:,:)
INTEGER :: seed(lseed)
INTEGER, ALLOCATABLE :: state(:)
! .. Executable Statements ..
WRITE (nout,*) 'G05RCF Example Program Results'
WRITE (nout,*)
FLUSH (nout)
! Skip heading in data file
READ (nin,*)
! Read in the base generator information and seed
READ (nin,*) genid, subid, seed(1)
! Initial call to initialiser to get size of STATE array
lstate = 0
ALLOCATE (state(lstate))
ifail = 0
CALL g05kff(genid,subid,seed,lseed,state,lstate,ifail)
! Reallocate STATE
DEALLOCATE (state)
ALLOCATE (state(lstate))
! Initialize the generator to a repeatable sequence
ifail = 0
CALL g05kff(genid,subid,seed,lseed,state,lstate,ifail)
! Read in sample size and number of dimensions
READ (nin,*) n, m
ldc = m
ldx = n
lr = m*(m+1) + 2
ALLOCATE (c(ldc,m),x(ldx,m),r(lr))
! Read in degrees of freedom
READ (nin,*) df
! Read in upper triangle portion of the covariance matrix
DO i = 1, m
READ (nin,*) c(i,i:m)
END DO
! Using a single call to G05RCF, so set up reference vector
! and generate values in one go
mode = 2
! Generate variates
ifail = 0
CALL g05rcf(mode,n,df,m,c,ldc,r,lr,state,x,ldx,ifail)
! Display the variates
ifail = 0
CALL x04caf('General',' ',n,m,x,ldx,'Variates',ifail)
END PROGRAM g05rcfe
